中国科学院植物种质创新与特色农业重点实验室草坪种质资源学学科组孙小艳博士在傅金民研究员的指导下,以课题组收集的100份来源于33个国家地区的高羊茅种质材料,在武汉夏季进行两轮高温处理,对每份基因型材料的生长速率(GR),草坪质量(TQ),存活率(SR),叶绿素含量(CHL),蒸腾速率(ET)进行实时采集,构建高羊茅耐高温功能性状数据库。并利用SSR标记技术完成了高羊茅群体结构分析和耐热性状关联分析。结果表明5个功能性状GR,TQ,SR,CHL和ET在高羊茅基因型间呈现显著遗传多样性。从200对高羊茅EST-SSR标记中筛选出100对多态性标记,连接上FAM、HEX、ROX和TAMRA等荧光基团,利用ABI 3730测序仪进行SSR位点检测,用Structure软件对100份高羊茅进行群体结构分析,用Structure 2.2软件的分群结果,在P<0.001的水平下将分1-3群的关联分析结构的Q值分别做关联分析。群体结构分析结果表明k=3时分群结果最佳。G1类群是最大的类群,包括91份高羊茅材料(包括23个商业品种和68份野生品种)。G2类群包括21个野生高羊茅材料,大部分来源于亚洲地区,G3类群最小,包含8份材料,其中5个野生高羊茅材料来源于非洲。基于Q + K混合模型,在两轮试验种分别鉴定有97 和 67个SSR位点与高羊茅耐热性状相关联(P < 0.01)。
该研究获得国家自然科学基金(Grant #: 31071822; 31470363)和国家863项目(Grant #: 2011AA100209-2)的支持。相关研究结果在植物学国际期刊BMC Plant Biology上在线发表(doi:10.1186/s12870-015-0494-5)。
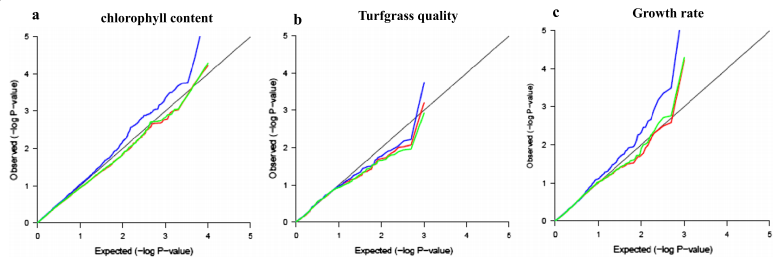
基于Q ,K ,Q +K 三模型的叶绿素含量、草坪质量和生长速率
Quantile-quantile plots 分析
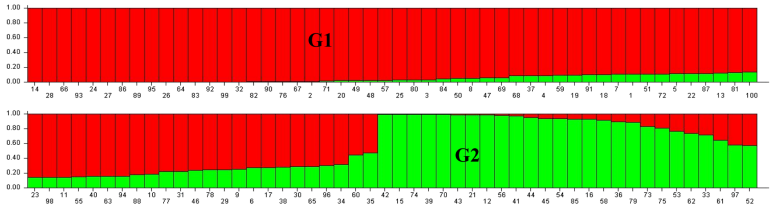
100份高羊茅种质材料的群体结构分析


