以往的分子系统发育研究往往基于单个或几个基因的序列。而在基因组时代,全基因组信息往往被利用研究分类群之间的进化关系。由于功能上的差异,不同的基因、基因家族有时会存在不同的进化历史,用不同基因家族的序列研究物种间的进化历史可能存在着准确度上的差异。例如,在植物中与胁迫抗病相关的R-gene进化上相对不保守,在不同类群中存在单独复制和丢失事件;而大多数管家基因(house keeping genes)在进化上并不发生复制,且序列上相对保守。
以基因树最简约法(Gene Tree Parsimony, GTP)为代表的系统基因组学(Phylogenomics)能够利用具有复杂的基因复制历史的基因家族数据来建立物种的系统发育树。GTP的方法主要寻找可以解释最少(基因复制)进化事件的最佳的物种系统进化树。然而,这种系统基因组学方法的准确性在不同进化模式的基因家族的背景下尚不了解。
中国科学院植物种质创新与特色农业重点实验室水生植物基因组学与遗传育种学科组助理研究员石涛利用多个被子植物的基因组序列,揭示了具备不同特征的基因家族进化历史对构建物种间的进化关系准确度的影响。该研究结果表明,基因家族的大小和基因家族在某个分类群中特异性扩增和缩小对GTP研究物种系统进化的准确性有强烈的影响,形成二项式曲线性(binomial)关系。根据这种进化模式对系统进化分析准确性影响的程度,可以量化GTP中的每次复制事件的生物学代价(biological cost),从而增加构建系统进化树的准确度。该研究发表在国际SCI期刊Molecular Phylogenetics and Evolution上。
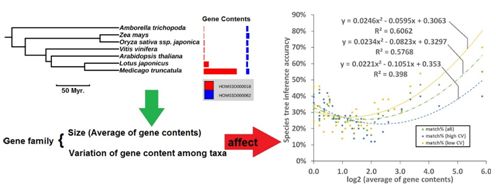
不同的基因家族的总体大小和在不同的分类群中的成员数有所不同,而这种差异直接影响着构建分类群的系统发育树的准确度


